
全舒
生物工程专业:博士生导师/硕士生导师
生物学专业:博士生导师/硕士生导师
生物与医药专业(生物工程领域):博士生导师/硕士生导师
生物反应器工程国家重点实验室,教授。2004年毕业于清华大学生物科学与技术系,获学士学位。同年赴美国密西根大学分子、细胞与发育生物学系攻读博士学位。2010年获得博士学位并于密西根大学及霍华德休斯医学研究所从事博士后研究工作。2014年加入350vip浦京集团,成立“蛋白质工程”课题组。长期聚焦蛋白质折叠这一重要科学问题,围绕分子伴侣及酶作用机制、蛋白质稳定性优化等展开基础及应用性基础研究。发展了筛选与鉴定分子伴侣的新策略,阐明了ATP非依赖型分子伴侣独特的作用机制;建立了蛋白质稳定性进化的系统性方法,在提升酶稳定性与减少淀粉样蛋白聚集方面展现了良好的应用前景。代表性成果以一作或通讯作者身份发表在Nature Structural & Molecular Biology,PNAS,Nature Communications,eLife等期刊上。先后获得上海市“浦江人才计划”(2014)、国家自然科学基金青年科学基金(2014)、我校 “青年英才引进与培育计划”(2015,2018)、我校杰出青年人才培育基金(2015)、国家自然科学基金面上项目(2017,2019,2022)、国家自然科学基金国际合作项目(2017)资助。
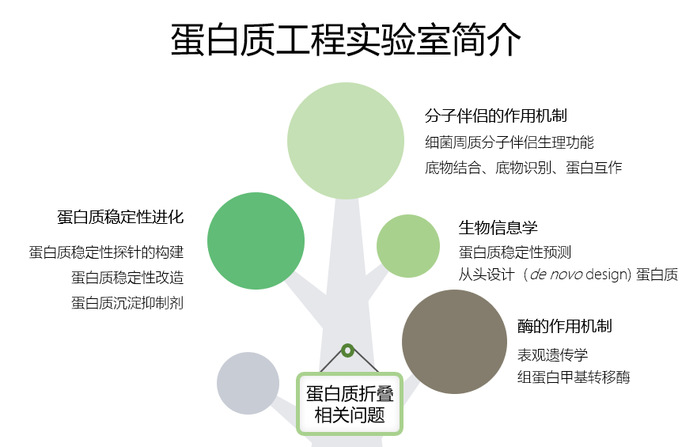
研究方向:
本课题组的研究兴趣为建立方法与手段改造蛋白质的稳定性,解析与蛋白质折叠密切相关的一类蛋白——分子伴侣蛋白的结构与作用机制,并利用所建立的蛋白质稳定性优化平台研究一些重要的基础科学问题,如参与表观遗传调控的一类组蛋白甲基转移酶的活化机制。我们将综合利用细菌遗传学、分子生物学、生物化学、生物物理、生物信息学与结构生物学等手段,开展如下几方面的研究工作:
1. 针对复杂蛋白质样品难以获得、不稳定的瓶颈问题,发展新型蛋白质体内稳定性检测探针,辅助以结构模拟与能量计算为基础的蛋白质理性设计,打造提升蛋白质稳定性的高通量筛选平台,研发新型高效异源蛋白质过表达体系,优化一些工业上、医药上迫切需要的蛋白质的稳定性。
2. 发现与鉴定新型分子伴侣蛋白,阐释其作用机理,并借助结构生物学、分子动力学模拟等手段研究分子伴侣蛋白和底物的相互作用、解析并表征体内蛋白质稳态网络,设计新型人工分子伴侣蛋白。
3. 筛选针对淀粉样蛋白的多肽抑制剂,为预防及治疗神经退行性疾病、二型糖尿病等蛋白质错误折叠引起的疾病提供新的思路。
4. 以提升组蛋白H3K4甲基转移酶家族的稳定性为切入点,解析结构灵活性与其催化活性间的关系,阐述其被其他蛋白质复合物激活的机制,为以此家族蛋白为靶点的药物设计提供新思路。
我们热忱欢迎有强烈好奇心与内驱力、期待自我提升、人际关系良好、心理素质过硬的新同学的加入!此外,我们正在大力探索人工智能前沿技术在蛋白质工程领域的应用,特别欢迎对此前沿交叉学科感兴趣、有一定编程基础的新同学加盟!
部分论文列表(#equal contribution, *通讯作者):
1. He W# (何为), Li X#, Xue H, Yang Y (杨园园), Mencius J (孟子钧), Bai L(白灵), Zhang J(张佳音), Xu J, Wu B*, Xue Y*, Quan S*. Insights into the client protein release mechanism of the ATP-independent chaperone Spy. Nature Communications 2022, 13: 2818.
2. Xue Z# (薛子孝), Pang Y# (庞永昊), Quan S*. Revisiting the functions of periplasmic chaperones in the quality control of the autotransporter Ag43 using a phenotypically homogeneous Escherichia coli strain. Biochemical and Biophysical Research Communications 2022, 581 (5): 37-43.
3. Ren C# (任畅), Zheng Y# (郑咏心), Liu C (刘春兰), Mencius J (孟子钧), Wu Z (吴治丽), Quan S*. Molecular characterization of an intrinsically disordered chaperone reveals net-charge regulation in chaperone action. Journal of Molecular Biology 2021, 434 (5): 167405.
4. He W# (何为), Yu G#, Li T (李天鹏), Bai L (白灵), Yang Y (杨园园), Xue Z (薛子孝), Pang Y (庞永昊), Reichmann D, Hiller S, He L*, Liu M*, Quan S*. Chaperone Spy protects outer membrane proteins from folding stress via dynamic complex formation. mBio2021, 12(5): e0213021.
5. Fan W# (范文轩,本科生), Mencius J# (孟子钧), Du W (杜文静,本科生), Fan H (范黄昀娴,本科生), Zhu H (朱泓锦,本科生), Wei D, Zhou M, Quan S*. Online bioinformatics teaching practice: comparison of popular docking programs using SARS-CoV-2 spike RBD-ACE2 complex as a benchmark. Biochemistry and Molecular Biology Education 2021, 49(6): 833-840.
6. Ren C (任畅), Wen X # (温欣), Mencius J # (孟子钧), Quan S*.An enzyme-based biosensor for monitoring and engineering protein stability in vivo. Proc Natl Acad Sci U S A 2021, 118(13):e2101618118.
7. Zheng Y (郑咏心), Huang Y #(黄银萍), Mencius J #(孟子钧), Li Y (黎彦璟), Zhao L, Luo W, Chen Y*, Quan S*, Distinct kinetic mechanisms of H3K4 methylation catalyzed by MLL3 and MLL4 core complexes. Journal of Biological Chemistry 2021, 296:100635.
8. He W (何为), Zhang J (张佳音), Sachsenhauser V, Wang L, Bardwell J, Quan S*, Increased surface charge in the protein chaperone Spy enhances its anti-aggregation activity. Journal of Biological Chemistry 2020, 295(42):14488-14500
9. Ruan A (阮青云), Ren C (任畅), Quan S*, Conversion of the molecular chaperone Spy into a novel fusion tag to enhance recombinant protein expression. Journal of Biotechnology 2020, 307:131-138
10. Ren C (任畅), Wen X (温欣), Mencius J (孟子钧), Quan S*. Selection and screening strategies in directed evolution to improve protein stability. Bioresources and bioprocessing 2019, 6, 53
11. Bazopoulou D, Knoefler D, Zheng Y (郑咏心), Ulrich K, Oleson B, Xie L, Kim M, Kaufmann A, Lee Y, Dou Y, Chen Y, Quan S, Jakob U*, Developmental ROS individualizes organismal stress resistance and lifespan. Nature 2019, 576:301–305.
12. Bai L (白灵), He W (何为), Li T (李天鹏), Yang C (杨翠婷), Zhuang Y, Quan S*, Chaperone-substrate interactions monitored via a robust TEM-1 β-lactamase fragment complementation assay. Biotechnology Letters. 2017 39(8): 1191-1199.
13. Quan S, Wang L, Petrotchenko EV, Makepeace K, Horowitz S, Yang J, Zhang Y, Borchers CH, Bardwell JCA*. Super spy variants implicate flexibility in chaperone action. eLife. 2014, 3:e01584.
14. Quan S, Tapley T, Koldewey P, Kirsch N, Ruane K, Pfizenmaier J, Shi R, Hofmann S, Foit L, Ren GP, Jakob U, Xu Z, Cygler M, Bardwell JCA*. Genetic selection designed to stabilize proteins uncovers a chaperone called Spy. Nature Structural & Molecular Biology. 2011, 18(3):262-269.
• Nature专栏评述: Powers ET & Balch WE. Protein folding: Protection from the outside. Nature 471(7336):42-43 (2011).
完整论文列表:
https://www.researchgate.net/profile/Shu-Quan/publications
教学与本科育人:
1. 主讲或参与:研究生全英文课程《Biocatalysis and Enzyme Technology》(2019 我校研究生教学成果二等奖),本科生全英文课程《Biochemistry》(上海市全英文示范课程、上海市一流本科课程认定),本科生全英文课程《Cell Biology》。
2. 第三届上海市大学生生命科学竞赛特等奖(2021)、全国大学生生命科学竞赛 (2021,科学探究类)一等奖。
实验室风采:





更多实验室情况请关注科学网博客:http://blog.sciencenet.cn/u/shuquan
联系方式:
上海市梅陇路130号350vip浦京集团,200237
办公室:实验十八楼1115室
电话:021-64253924
E-mail: shuquan@ecust.edu.cn